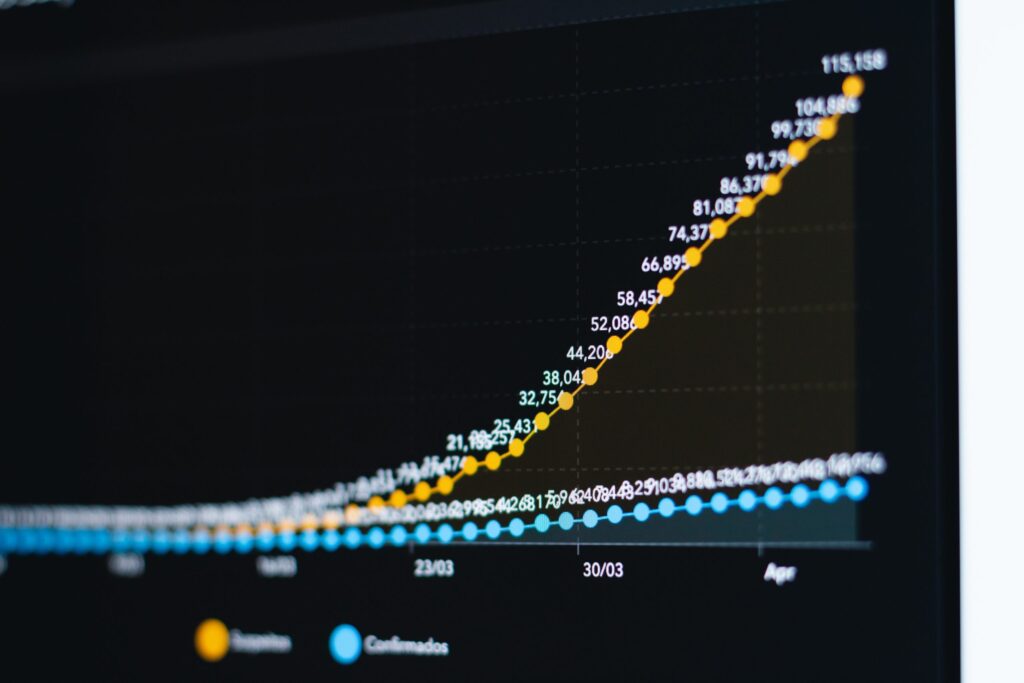
จากสถานการณ์โรคระบาด COVID-19 ที่เกิดขึ้นในปัจจุบัน หลายคนคงจะเห็นกราฟแสดงจำนวนผู้ติดเชื้อมากมายหลายรูปแบบผ่านทางอินเตอร์เน็ตหรือทางโทรทัศน์ แต่อาจจะยังไม่ทราบว่า เราสามารถสร้างกราฟสวย ๆ เหล่านั้นไว้เพื่อติดตามสถานการณ์หรือวิเคราะห์ได้ด้วยตนเอง โดยในบทความนี้เราจะมาสร้างกราฟแสดงจำนวนผู้ติดเชื้อ COVID-19 ในประเทศไทยผ่านโปรแกรม R Studio กัน บอกเลยว่า มันง่ายมาก ๆ และหากใครที่ยังไม่รู้จักโปรแกรม R เรามาเริ่มทำความรู้จักกันเลยดีกว่าครับ
ภาษา R เป็นภาษาทางคอมพิวเตอร์ที่นิยมใช้อย่างกว้างขวางมากในหมู่นักวิเคราะห์ข้อมูลทางสถิติและมีแนวโน้มที่จะได้รับความนิยมเพิ่มมากขึ้นเรื่อย ๆ ทั้งนี้ เนื่องจากการที่ทุกคนสามารถใช้ R ได้ฟรีโดยไม่ต้องเสียค่าใช้จ่าย ภาษา R จึงได้รับความนิยมเป็นอย่างมากจากทั้งวงการวิชาการ และภาคเอกชน อีกทั้ง ผลการวิเคราะห์ทางสถิติด้วย R นั้น ยังได้รับการยอมรับจากนานาชาติ และยังสามารถใช้งานได้ทั้งในระบบปฏิบัติการ Window PC, Mac และ Linux 1.2.4
ปกติแล้วเราเขียนภาษา R บนไหนกัน?
โปรแกรมที่ช่วยให้เราเขียนภาษา R ได้ง่ายนั้นก็คือ R Studio โดยผู้ใช้สามารถปรับแต่ง UI ให้เหมาะกับการใช้งานของตัวเอง แถมยังมีความยืดหยุ่นสูงอีกด้วย

รูปที่ 1 โลโก้ของโปรแกรม R และ R Studio
สำหรับผู้อ่านที่อยากจะลองทำตาม tutorial ในบทความนี้ ท่านจะต้องลงโปรแกรม R และ RStudio ก่อนนะครับ โดยท่านสามารถดาวน์โหลดทั้งสองโปรแกรมได้ที่ลิ้งนี้เลย
R Program -> https://cran.r-project.org/ (เลือกลงได้เลย: Windows | Mac | Linux)
R Studio -> https://www.rstudio.com/
ข้อมูลผู้ติดเชื้อ COVID-19 ในประเทศไทย
เราจะทำการดึงข้อมูลจากศูนย์กลางการให้บริการข้อมูลเปิดภาครัฐ (Open Government Data) (https://www.data.go.th/dataset/covid-19-daily) เราสามารถดึงข้อมูลจากเว็บไซต์นี้ออกมาได้หลายรูปแบบเลยครับ แต่เพื่อให้ผู้ที่พึ่งรู้จักกับภาษา R ได้เข้าใจง่ายขึ้น เราจะดึงออกมาในรูปแบบ Excel กันนะครับ
ก่อนอื่นก็เข้าไปที่เว็บไซต์ข้างต้น แล้วโหลดข้อมูลเข้ามาไว้ที่เครื่องของตัวเองกันได้เลยครับ
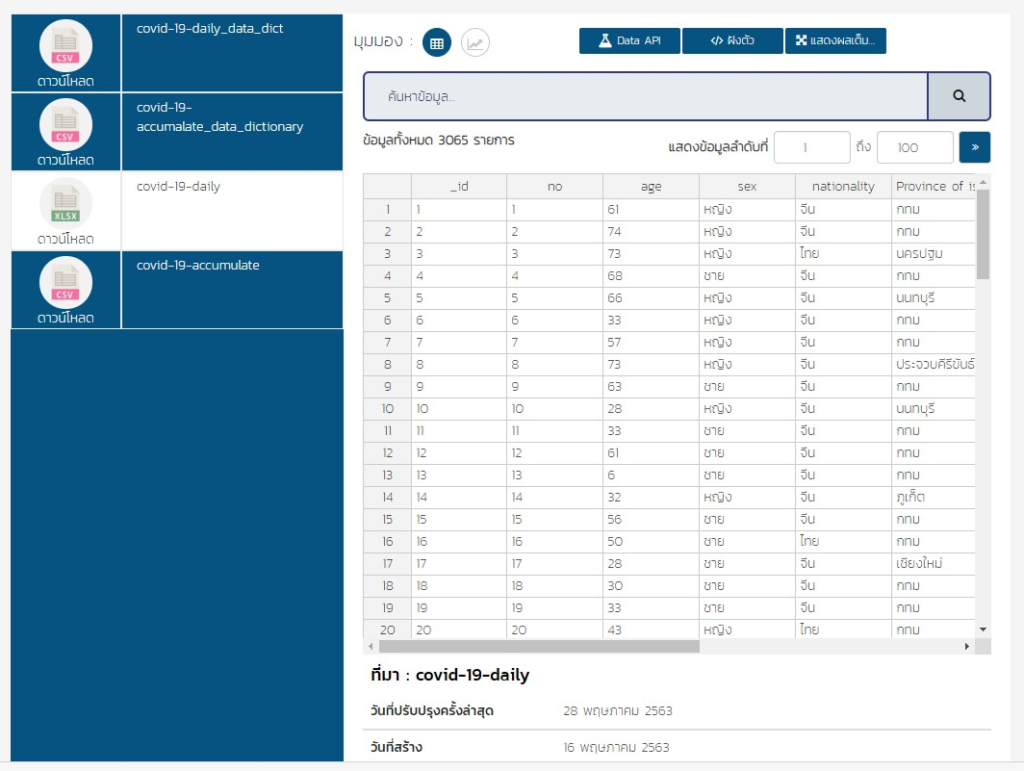
หลังจากที่ผู้อ่านได้ข้อมูลและลงโปรแกรมเป็นที่เรียบร้อยแล้ว เรามาเริ่มสร้างกราฟกันเลยดีกว่าครับ
นี่คือหน้าตาของโปรแกรม R Studio ครับ น่าใช้มากเลยใช่ไหมครับ หากใครไม่ชอบ Dark mode หรือ font ในการเขียนโค้ด ก็สามารถเปลี่ยนตามที่ชอบได้เลยครับ
เริ่มแรกเราจะต้องติดตั้งและเรียกใช้ Package ที่จำเป็นจะต้องใช้ในการสร้างกราฟ โดยผมได้รวมมาให้แล้วครับ ให้ทุกคนนำโค้ดด้านล่างวางบน R script จากนั้นก็กด run กันได้เลยครับ
> install.packages("tidyverse","ggplot2","dplyr","hrbrthemes","ggpubr")
> library(tidyverse)
> library(ggplot2)
> library(dplyr)
> library(scales)
> library(hrbrthemes)
> library(readxl)
> library(ggpubr)เมื่อติดตั้ง package และทำการเรียกใช้ package ทั้งหมดนี้ด้วยคำสั่ง library() กันเสร็จแล้ว ขั้นตอนแรก ผมอยากให้ทุกคน import ข้อมูลเข้ามาในโปรแกรม โดยการ click import จากเมนูหรือ run ตาม script แค่เปลี่ยน Location ที่ข้อมูลวางอยู่ให้ตรงกับที่อยู่ของไฟล์ในเครื่องคอมพิวเตอร์ของคุณเท่านั้นเองครับ
> Covid_Th <- read_excel("C:/…………/20200528_pm.xlsx")ผมได้เก็บข้อมููลไว้ในชื่อ Covid_Th
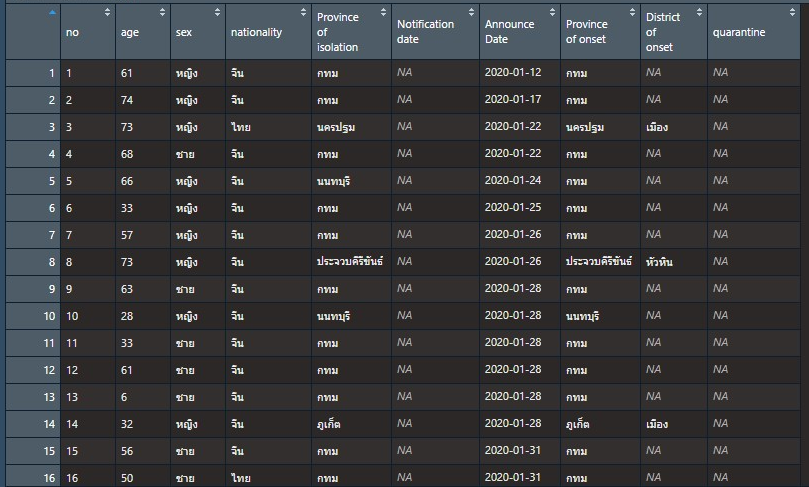
หลังจากที่เราได้ข้อมูลมาแล้ว เราจะทำการเปลี่ยนประเภทของคอลัมน์ Announce Date ให้อยู่ในรูปของวันที่ (Date Format) ที่โปรแกรม R เข้าใจและสามารถนำมาสร้างกราฟได้อย่างถูกต้อง
> Covid_Th$`Announce Date` <- as.Date(Covid_Th$`Announce Date`, "%m/%d/%Y")วิธีตรวจสอบก็คือ
> class(Covid_Th$`Announce Date`)
[1] "Date"จะเห็นว่า คอลัมน์นี้เป็นข้อมูลประเภทวันที่เรียบร้อยแล้ว
Package ที่เราจะนำมาสร้างกราฟคือ ggplot2 Package นี้มีประโยชน์มาก ท่านสามารถที่จะนำข้อมูลมาสร้างกราฟได้ละเอียด ตามที่ต้องการ Package ggplot2 นี้มีคนใช้และพัฒนาอยู่เป็นจำนวนมาก ทำให้เราสามารถเล่นกับ package นี้ได้หลากหลายรูปแบบมากเลยครับ เรามาเริ่มกันเลยดีกว่าครับ
ผมจะสร้าง Dataset ใหม่ขึ้นมา เพื่อนับจำนวนผู้ป่วย COVID-19 ในแต่ละวันชื่อว่า Covid_Th_group
> Covid_Th_group <- Covid_Th %>%
group_by(`Announce Date`) %>%
summarise(count = n())เมื่อผมนำ Covid_Th_group ไปพลอต จะได้ผลลัพธ์ตามด้านล่างนี้ครับ
> Covid_Th_Plot <- ggplot(data = Covid_Th_group, aes(x = `Announce Date`, y = `count`)) +
geom_line(size = 0.8,color = "#00AFBB")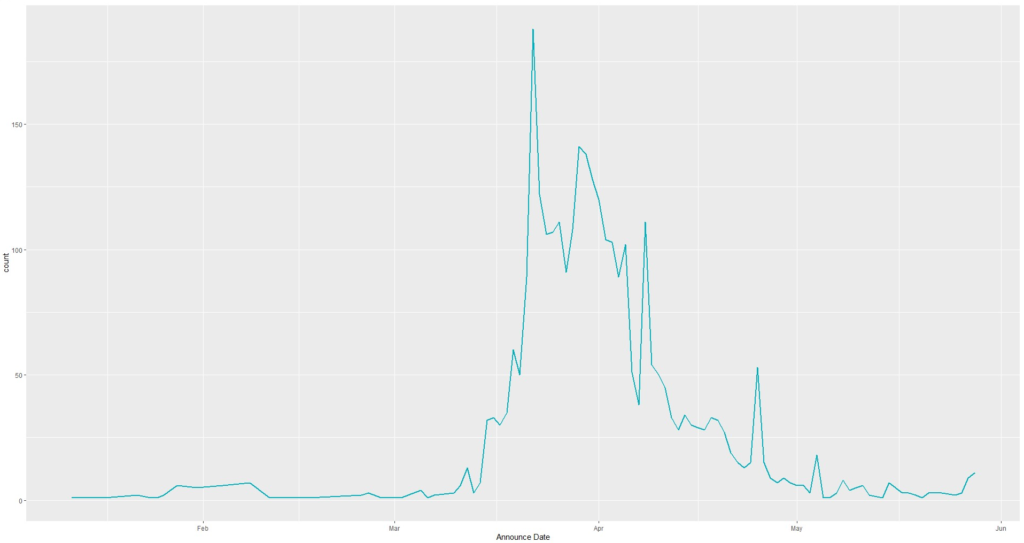
ผมได้กราฟที่ต้องการมาแล้ว ง่ายมากเลยใช่ไหมครับ แต่อย่างที่ผมได้บอกไว้ข้างต้นว่า เราสามารถปรับแต่งกราฟตามที่เราชอบได้เลยรวมถึงการใส่คำบรรยายเพื่อให้เข้าใจกราฟนี้มากขึ้น ตัวอย่างโค้ดที่ใช้ในการสร้างกราฟตามสไตล์ของผมนั้นอยู่ด้านล่างนี้เลยครับ
> Covid_Th_Plot <- Covid_Th_Plot +
geom_point() +
labs(title="จำนวนผู้ติดเชื้อ Covid-19 ในประเทศไทยรายวัน",
caption ="Source: https://data.go.th/dataset/covid-19-daily",x = "Date", y = "Number") +
xlab("") +
scale_x_date(labels = date_format("%Y %b %d"),date_breaks = "5 day") +
theme_ipsum() +
theme(axis.text.x=element_text(angle=60, hjust=1),
panel.grid.major.x = element_blank(),
panel.grid.minor.x = element_blank())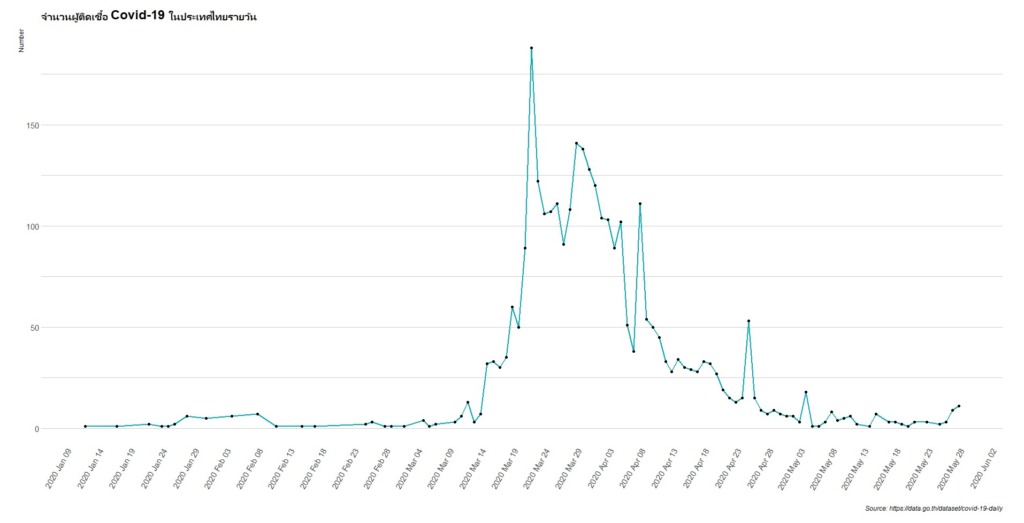
นอกจากนี้ เรายังสามารถแบ่งกลุ่มการพลอตกราฟตามคอลัมน์ที่ต้องการได้ด้วยนะครับ ผมจะพลอตกราฟแยกตามเพศของผู้ที่ติดเชื้อนะครับ
> Covid_Th_Plot2 <- ggplot(data = Covid_Th %>% group_by(`Announce Date`,`sex`) %>%
summarise(count = n()), aes(x = `Announce Date`, y = `count`)) +
geom_line(aes(col = sex),size = 0.8) +
scale_color_manual(values = c("#00AFBB", "#E7B800")) +
scale_fill_manual(values = c("#00AFBB", "#E7B800")) +
labs(title="จำนวนผู้ติดเชื้อ Covid-19 ในประเทศแบ่งตามเพศของผู้ติดเชื้อรายวัน",
caption ="Source: https://data.go.th/dataset/covid-19-daily",
x = "Date", y = "Number") +
xlab("") +
scale_x_date(labels = date_format("%Y %b %d"),date_breaks = "5 day") +
theme_ipsum() +
theme(axis.text.x=element_text(angle=60, hjust=1),
panel.grid.major.x = element_blank(),
panel.grid.minor.x = element_blank())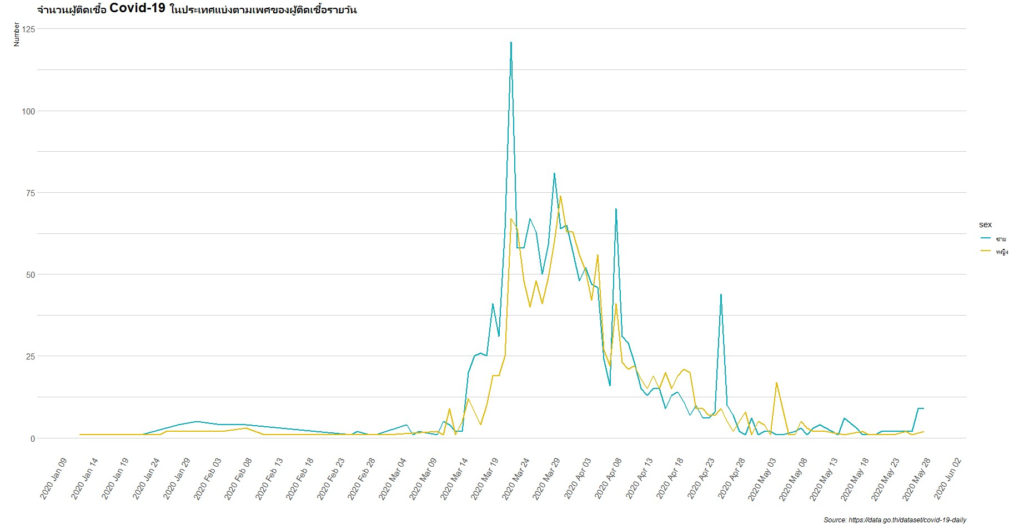
หากเราอยากรวมทั้งสองกราฟไว้บนหน้าจอเดียวกัน เพื่อจะแสดงกราฟทั้งสองรูปพร้อมกันก็ทำได้เช่นกันครับ
> ggarrange(Covid_Th_Plot, Covid_Th_Plot2, nrow = 2)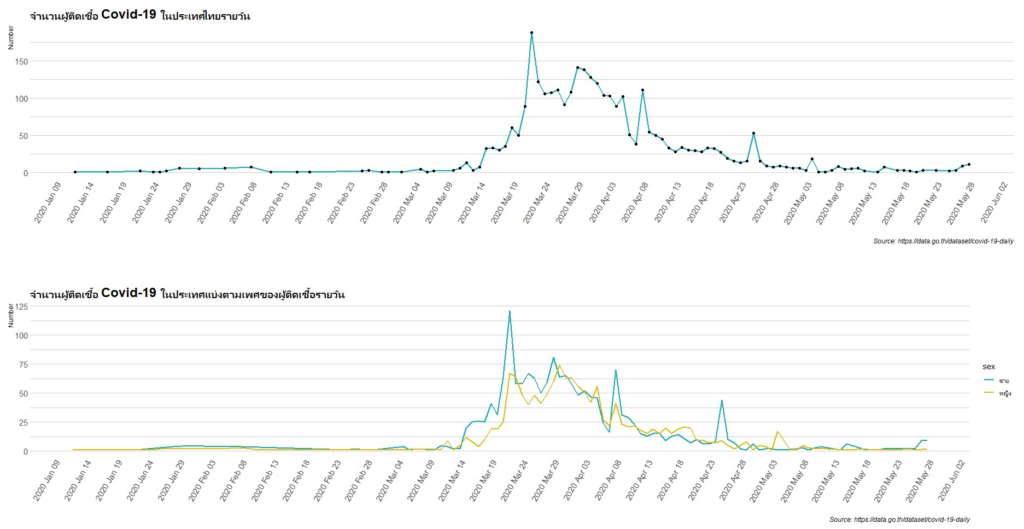
นอกจากกราฟเส้นที่ได้แสดงไปแล้วนั้น Package ggplot2 ยังได้ถูกออกแบบมาให้เราสามารถสร้างกราฟชนิดอื่นได้อีกด้วย หากอยากเปรียบเทียบเพศในแต่ละวันผ่านกราฟแท่ง ก็สามารถทำได้ด้วยการแก้ code “geom_line” เป็น “geom_bar” ตามด้านล่างเลยครับ
> ggplot(Covid_Th, aes(`Announce Date`)) +
geom_bar(aes(fill = sex), position = position_stack(reverse = FALSE)) +
scale_color_manual(values = c("#00AFBB", "#E7B800")) +
scale_fill_manual(values = c("#00AFBB", "#E7B800")) +
labs(title="เปรียบเทียบจำนวนผู้ติดเชื้อ Covid-19 รายวันตามเพศสภาพ",
caption ="Source: https://data.go.th/dataset/covid-19-daily",
x = "Date", y = "Number") +
xlab("") +
scale_x_date(labels = date_format("%Y %b %d"),date_breaks = "5 day") +
theme_ipsum() +
theme(axis.text.x=element_text(angle=60, hjust=1),
panel.grid.major.x = element_blank(),
panel.grid.minor.x = element_blank())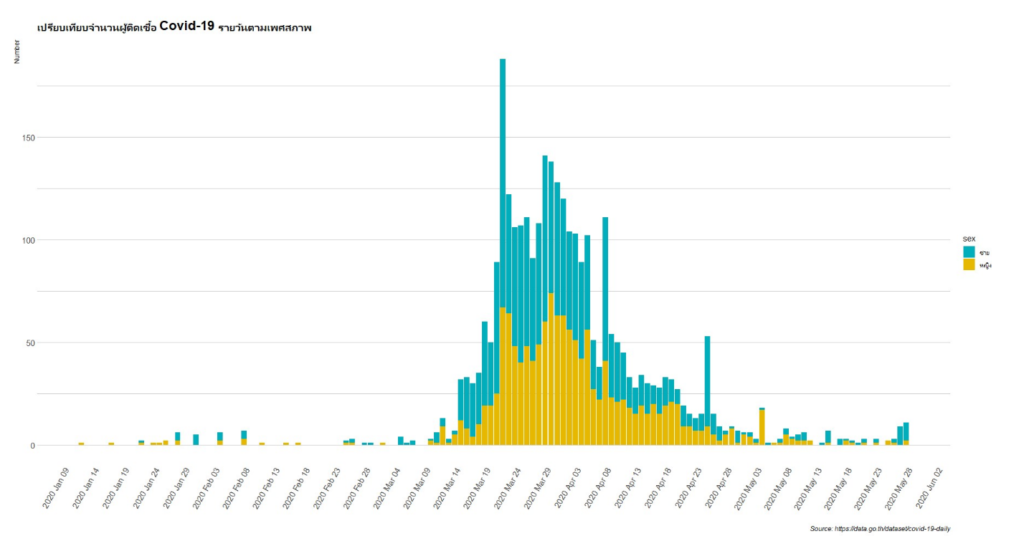
กราฟแท่งที่แสดงอยู่ด้านบนนี้ อาจจะทำให้ผู้วิเคราะห์มองภาพได้ง่ายขึ้นกว่ากราฟเส้นในการเปรียบเทียบจำนวนผู้ป่วยตามเพศชายและหญิงในแต่ละวันครับ
หรือในกรณีที่เราอยากทราบว่า จังหวัดใดมีผู้ติดเชื้อเท่าไหร่ เราก็สามารถเปรียบเทียบจำนวนของผู้ติดเชื้อรายจังหวัด โดยแก้ไขที่ group เปลี่ยนจากวันที่เป็นจังหวัดแทนครับ
> ggplot(data = Covid_Th , aes(x =`Province of onset`)) +
geom_bar(aes(fill = `sex`)) +
labs(title="เปรียบเทียบจำนวนผู้ติดเชื้อ Covid-19 ตามจังหวัด",
caption ="Source: https://data.go.th/dataset/covid-19-daily",
x = "Province of onset", y = "Number") +
xlab("") +
theme_ipsum() +
theme(axis.text.x=element_text(angle=60, hjust=1),
panel.grid.major.x = element_blank(),
panel.grid.minor.x = element_blank()) 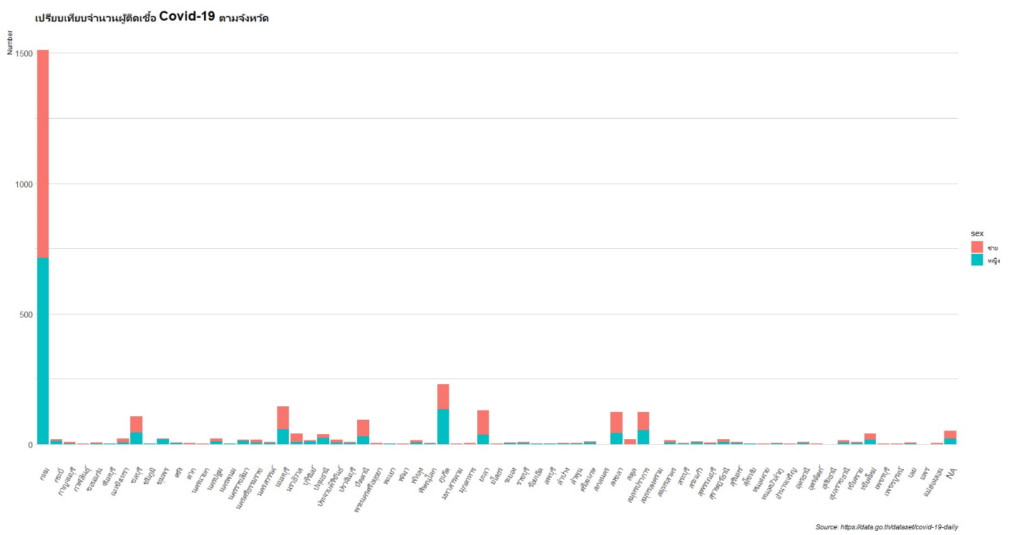
จะเห็นได้ว่า กรุงเทพเป็นจังหวัดที่มีจำนวนผู้ติดเชื้อมากที่สุดเมื่อเปรียบเทียบกับจังหวัดอื่น โดยสัดส่วนของเพศชายและเพศหญิงที่ติดเชื้อในกรุงเทพนั้นก็ใกล้เคียงกันมาก หากอยากทราบเพิ่มเติมว่า สัดส่วนการติดเชื้อของเพศชายและเพศหญิงต่างกันกี่เปอร์เซ็นต์ เราก็สามารถสร้างกราฟวงกลมเพื่อเปรียบเทียบเพิ่มเติมได้อีกนะครับ
> Covid_Th_group2 <- Covid_Th %>%
group_by(`sex`) %>%
summarise(count = n())
> ggplot(Covid_Th_group2, aes(x="", y=count, fill=sex)) +
geom_bar(stat="identity", width=1) +
coord_polar("y", start=0) +
theme_minimal() +
theme(axis.text.x=element_blank()) +
geom_text(aes(label = paste0(round(count*100/sum(count)), "%")), position = position_stack(vjust = 0.5),size=8) +
labs(x = NULL, y = NULL, fill = NULL, title = "สัดส่วนเพศของจำนวนผู้ติดเชื้อ Covid-19 ในประเทศไทย")+
theme_classic() +
theme(axis.line = element_blank(),
axis.text = element_blank(),
axis.ticks = element_blank(),
plot.title = element_text(hjust = 0.5,size = 25),
legend.text = element_text(size = 20))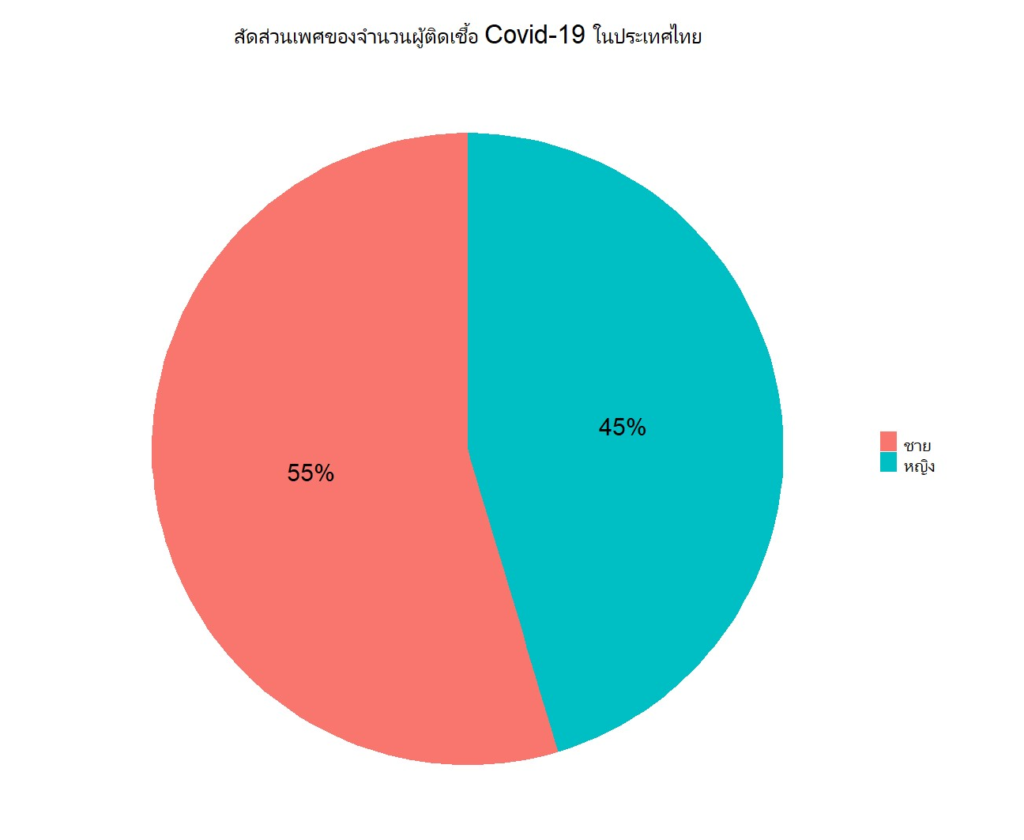
จากบทความข้างต้น จะเห็นได้ว่า เราสามารถสร้างกราฟสวย ๆ แสดงจำนวนผู้ติดเชื้อ COVID-19 ได้ไม่ยากจากการใช้โปรแกรม R Studio ซึ่งเป็นโปรแกรมฟรี ไม่มีค่าใช้จ่าย ผู้อ่านสามารถติดตั้งและใช้งาน package ggplot2 ภายในโปรแกรม R Studio ได้เลยครับ ซึ่งการใช้ package ต่าง ๆ จะทำให้เราเขียนภาษา R เพื่อทำการวิเคราะห์ข้อมูลได้ง่ายขึ้นมาก สำหรับ package ggplot2 นี้ถือเป็น package ยอดนิยมในการสร้างกราฟของเหล่าบรรดาสาวก R อีกด้วยครับ ในบทความนี้ผมได้นำข้อมูลผู้ติดเชื้อ COVID-19 มาเป็นตัวอย่างในการทำ Visualization เพื่อเป็นไอเดียให้ทุกคนสามารถนำไปประยุกต์กับข้อมูลที่สนใจหรือทำอยู่ในปัจจุบันได้ง่ายขึ้นครับ
Data Scientist
Government Big Data Institute: GBDi
- Yuranan Jamjureehttps://temp.bdi.or.th/en/author/yuranan-ja/
- Yuranan Jamjureehttps://temp.bdi.or.th/en/author/yuranan-ja/18 November 2020